Single cell RNA-Seq
Ofrecemos el análisis bioinformático de datos NGS para células individuales
Portada » Single cell RNA-Seq
Estudio de la expresión génica a nivel celular
El análisis Single cell RNA-Seq permite analizar el transcriptoma de cada célula de manera individual y puede revelar procesos biológicos y mecanismos moleculares específicos que podrían no ser detectados mediante técnicas de secuenciación de ARN a nivel poblacional o de tejido.
El análisis bioinformático que llevamos a cabo en Dreamgenics incluye:
- Control de calidad de la secuenciación y mapeado.
- Alineamiento de lecturas frente a genoma de referencia.
- Cuantificación de la expresión génica a nivel de célula única.
- Reducción de la dimensionalidad de los datos.
- Clustering o división de las células en distintos grupos y subgrupos.
- Análisis de la expresión génica diferencial.
- Estudio del patrón evolutivo o de maduración celular (Velocity o Análisis de Trayectorias).
- Estudio de enriquecimiento de ontologías génicas y pathways.
Archivos de entrada
Podemos llevar a cabo el análisis bioinformático a partir de:
- FASTQ de Illumina
- Tecnología 10X
Puedes enviarnos tus archivos crudos en un disco duro o compartirlos a través de un FTP.
Resultados en Genome One Reports
Los resultados de todos los análisis dan lugar a gráficos interactivos que se pueden explorar en nuestra plataforma Genome One Reports y descargarse en PDF para su uso en publicaciones científicas.
- Tendrás acceso al análisis de calidad de las secuencias obtenidas
- Tendrás acceso a los resultados del análisis de Cell Ranger
- Gráficos PCA plot, Cell Clustering, Trajectory Analisis, RNA Velocity, Cell Chat, etc. en función del paquete elegido
- Enriquecimiento de rutas y términos Gene Ontologies (WikiPathways, GO-MF, GO-CC y GO-BP)
- Todas las tablas y figuras son fácilmente exportables, listas para ser utilizadas en publicaciones científicas
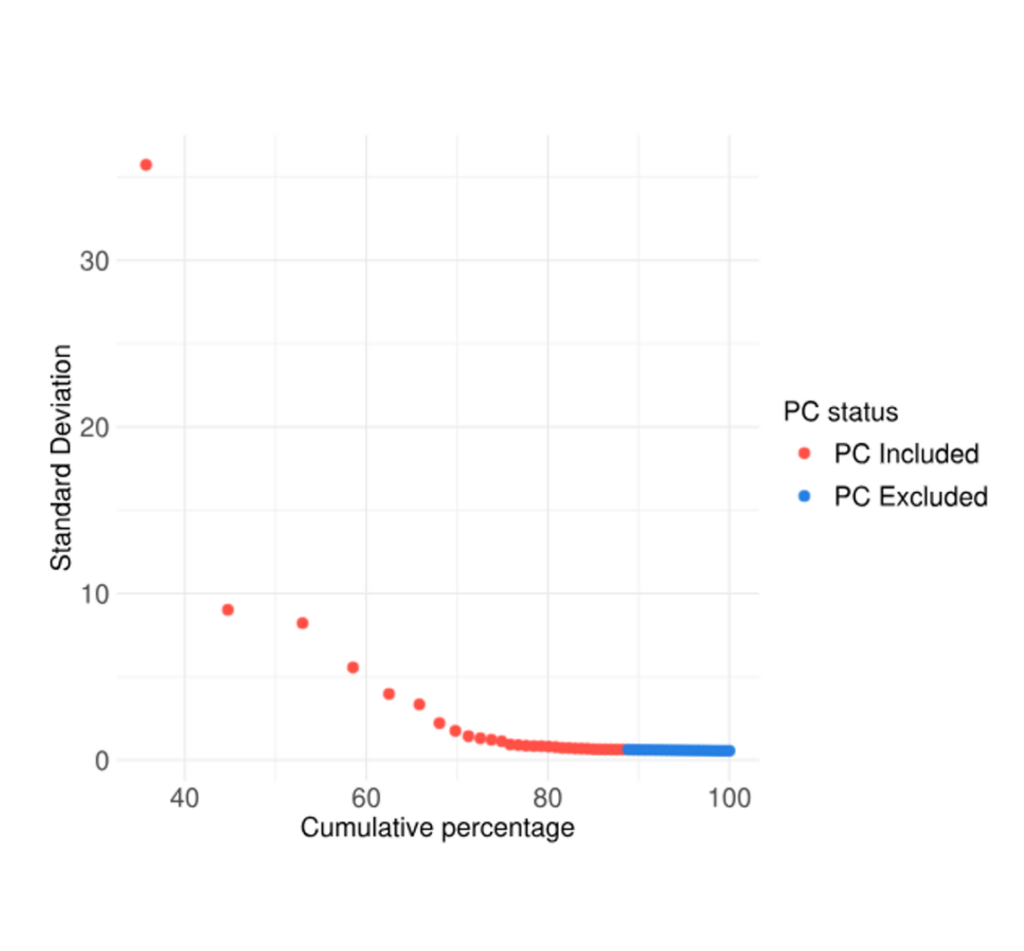
PCA Plot
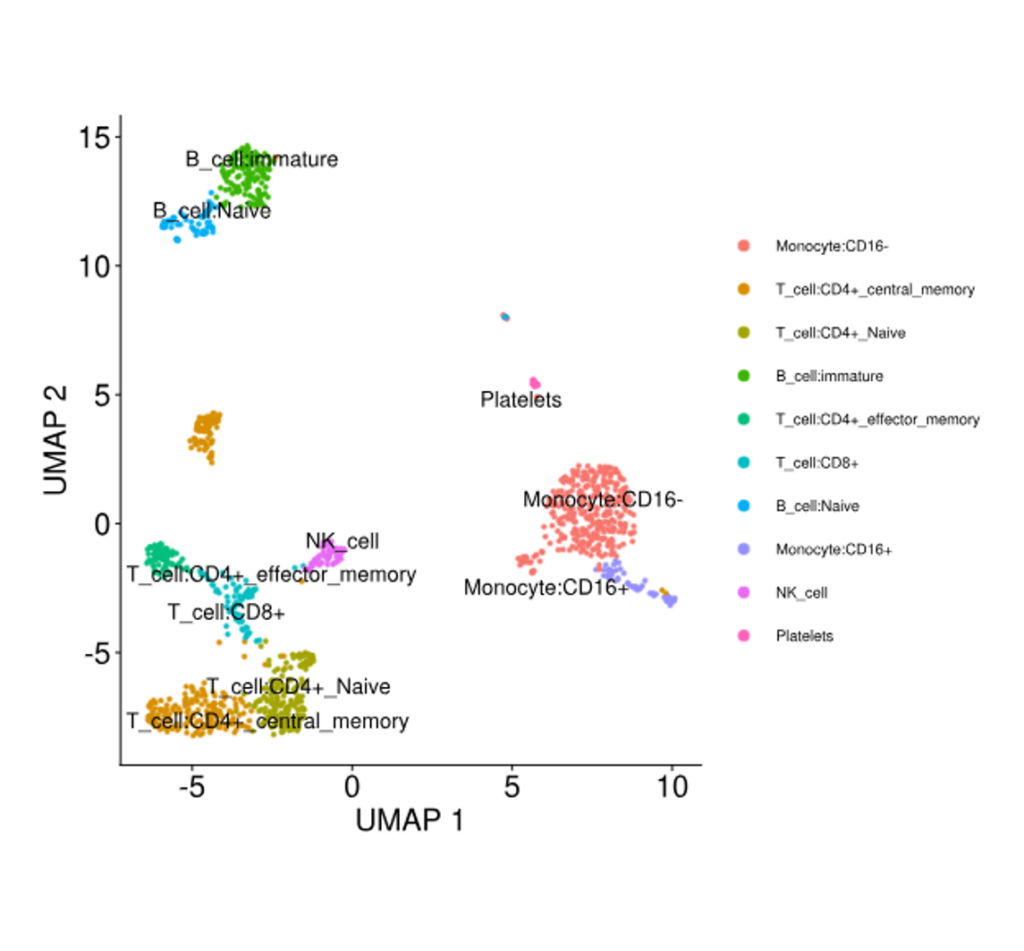
Cell Clustering
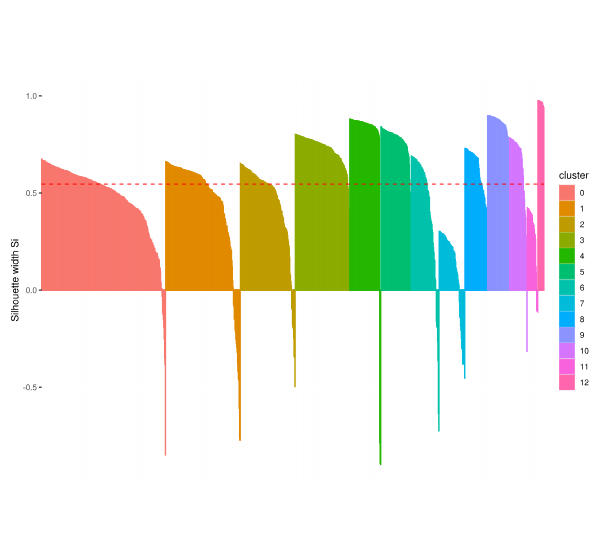
Cluster Evaluation
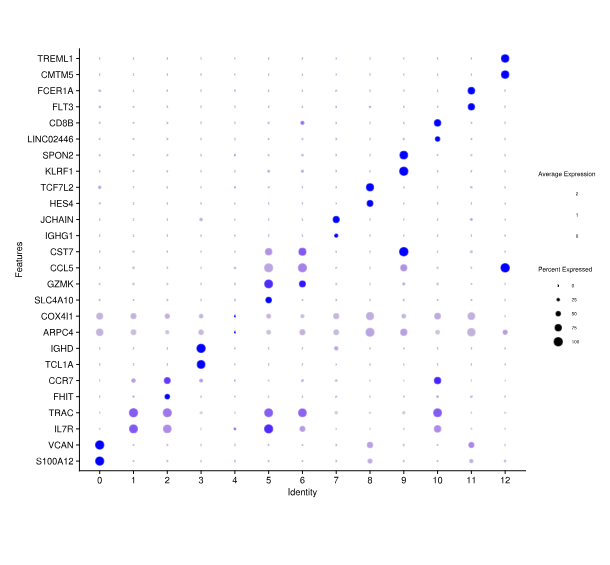
Differentially Expressed Genes
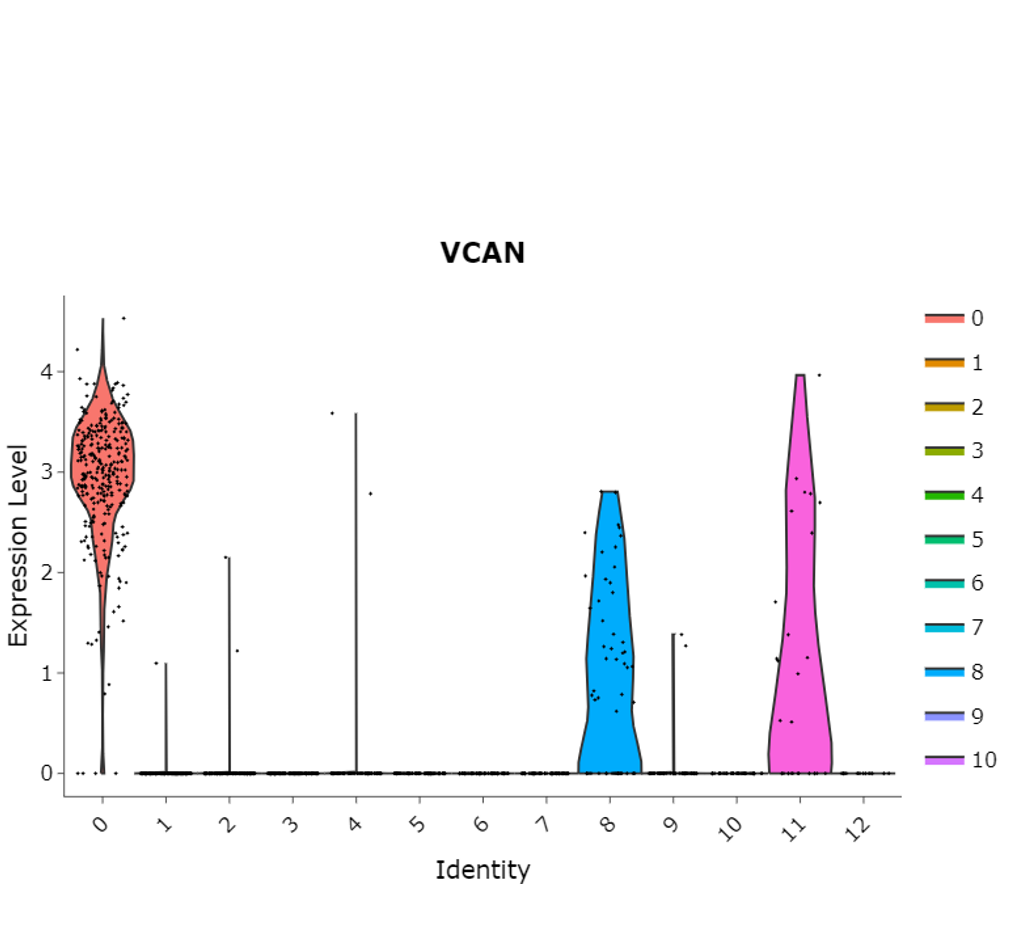
Violin Plot
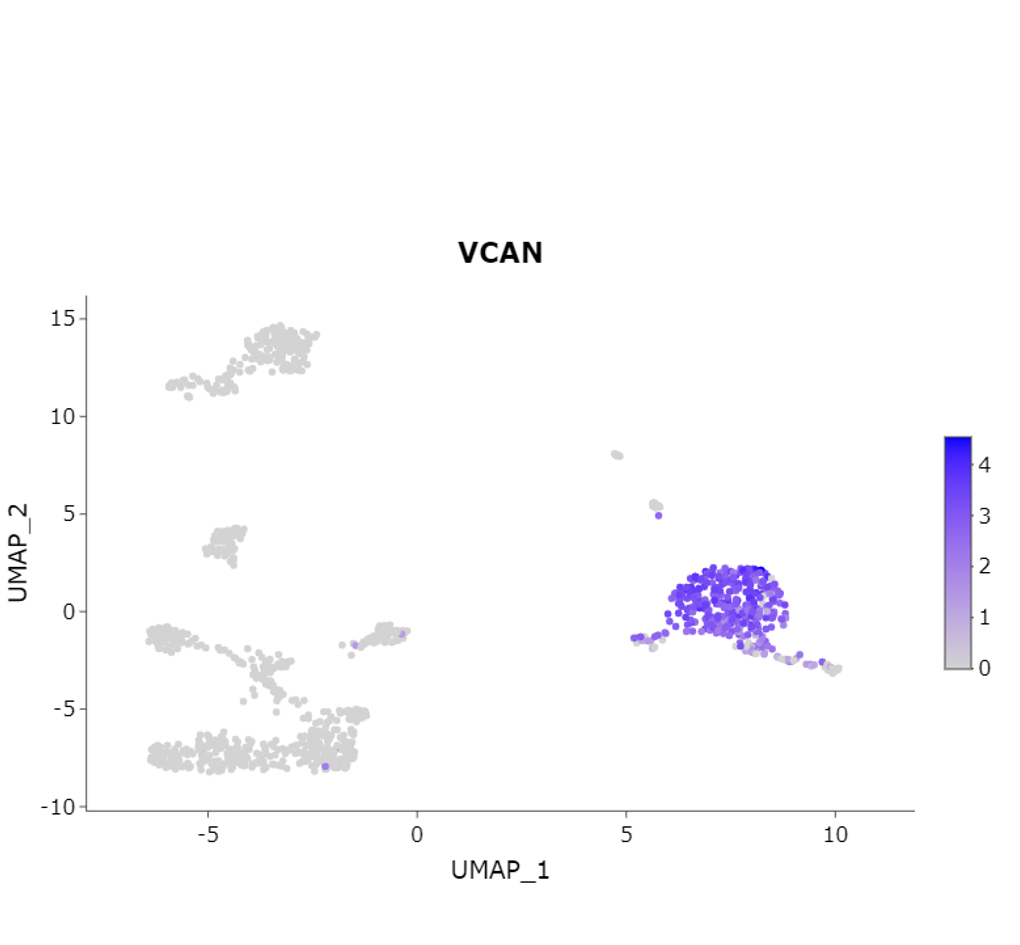
Feature Plot
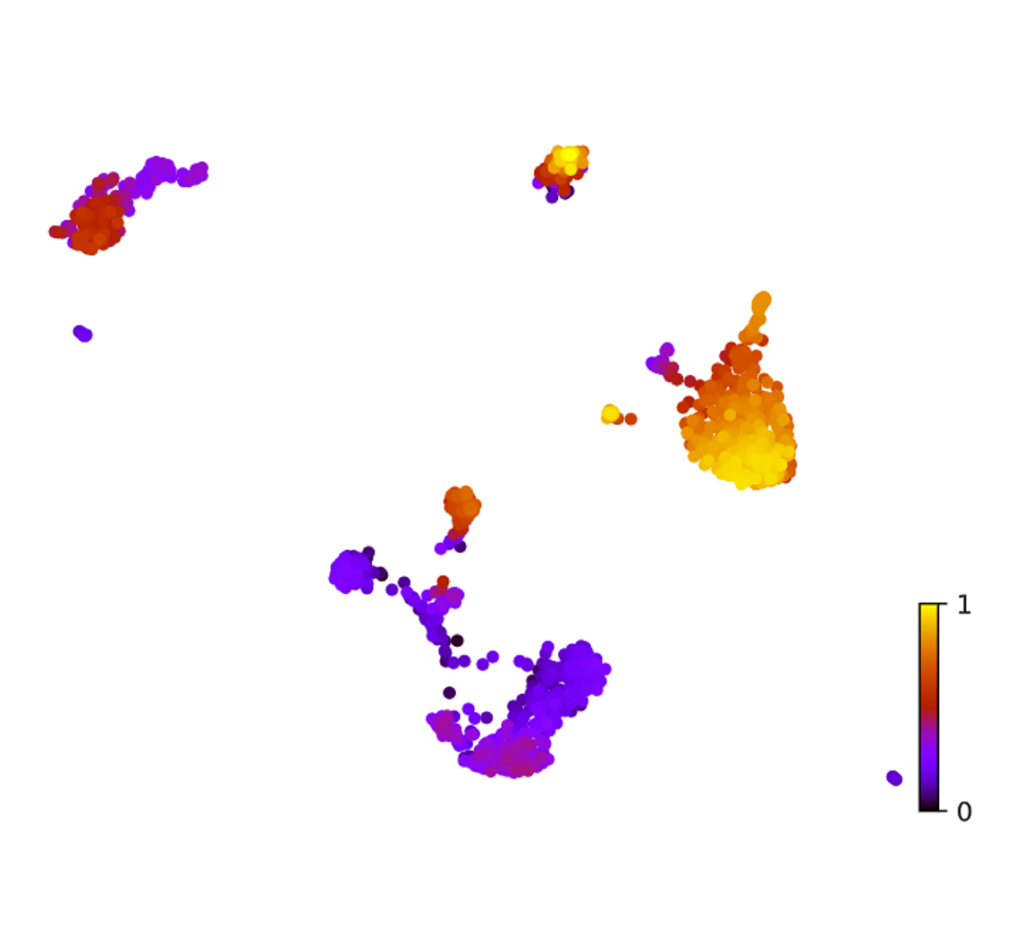
RNA Velocity
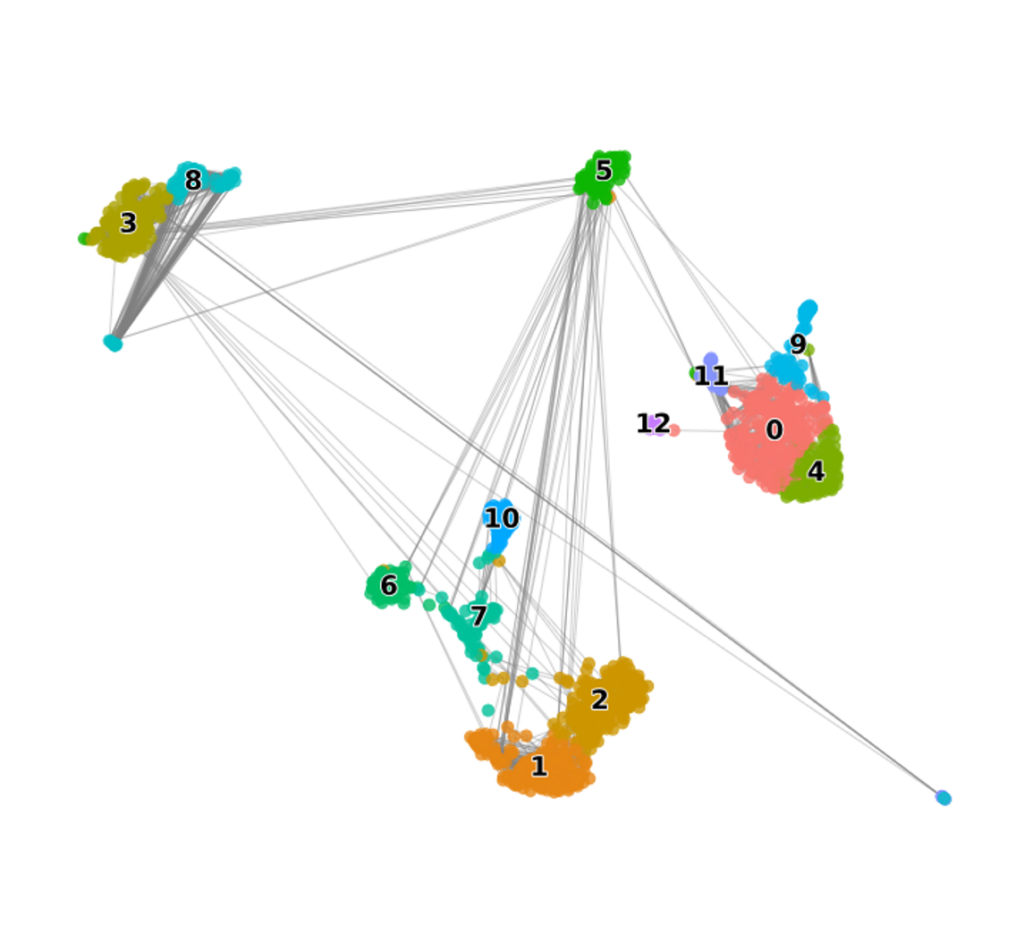
Neighbor Graph
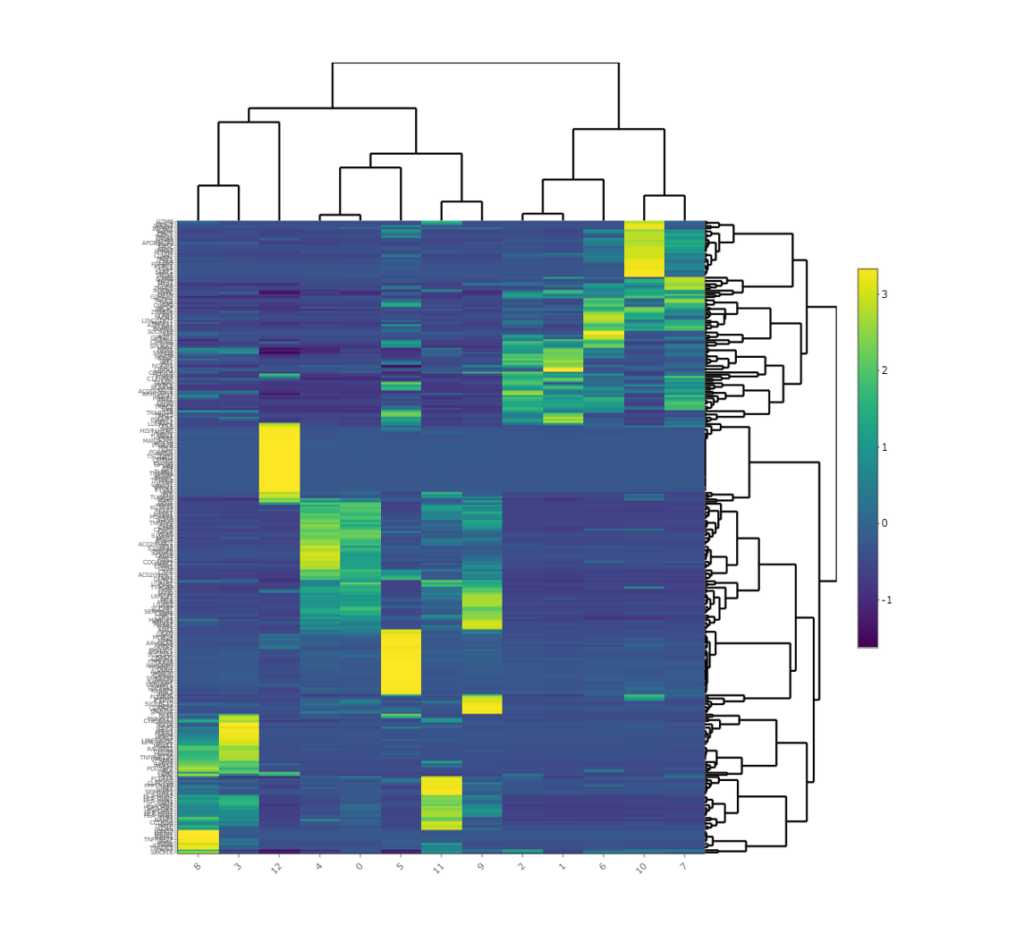
Heatmap
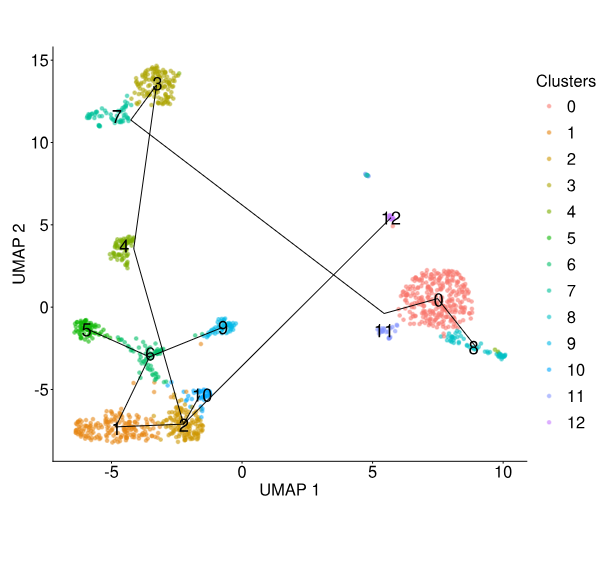
Trajectory Analysis
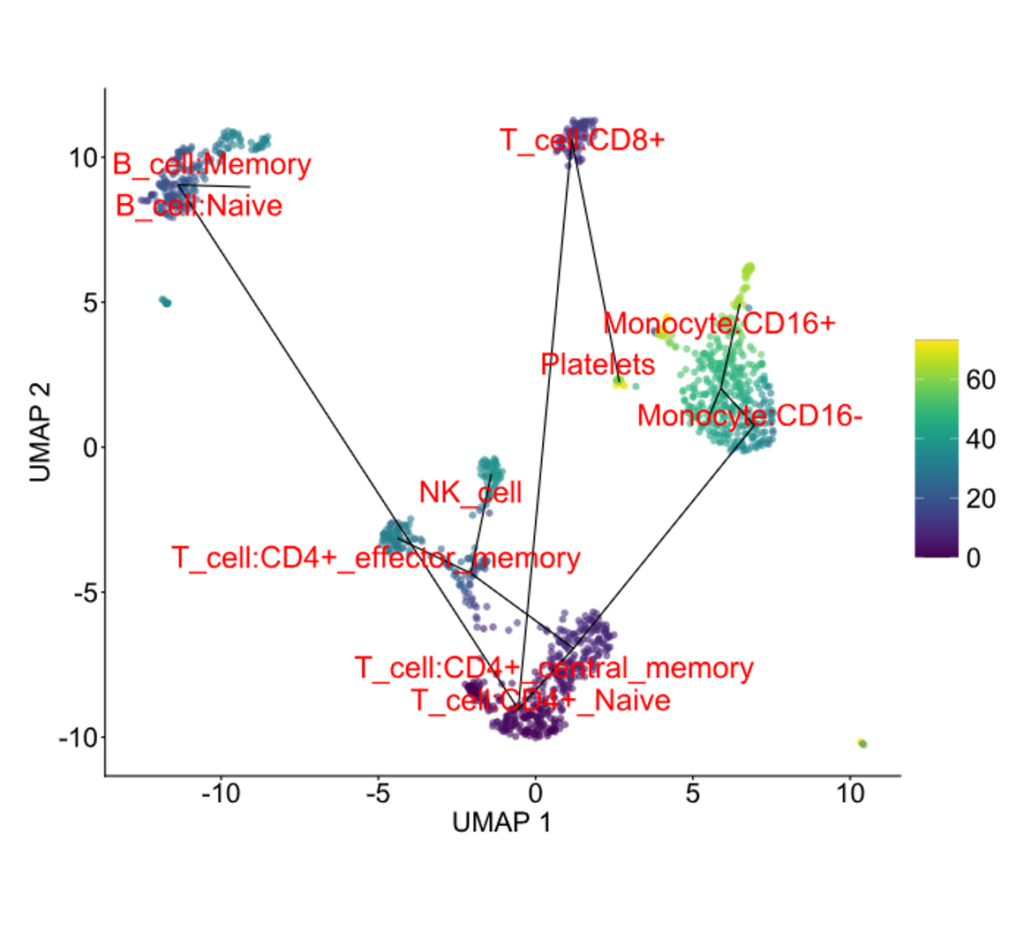
Pseudotime trajectory
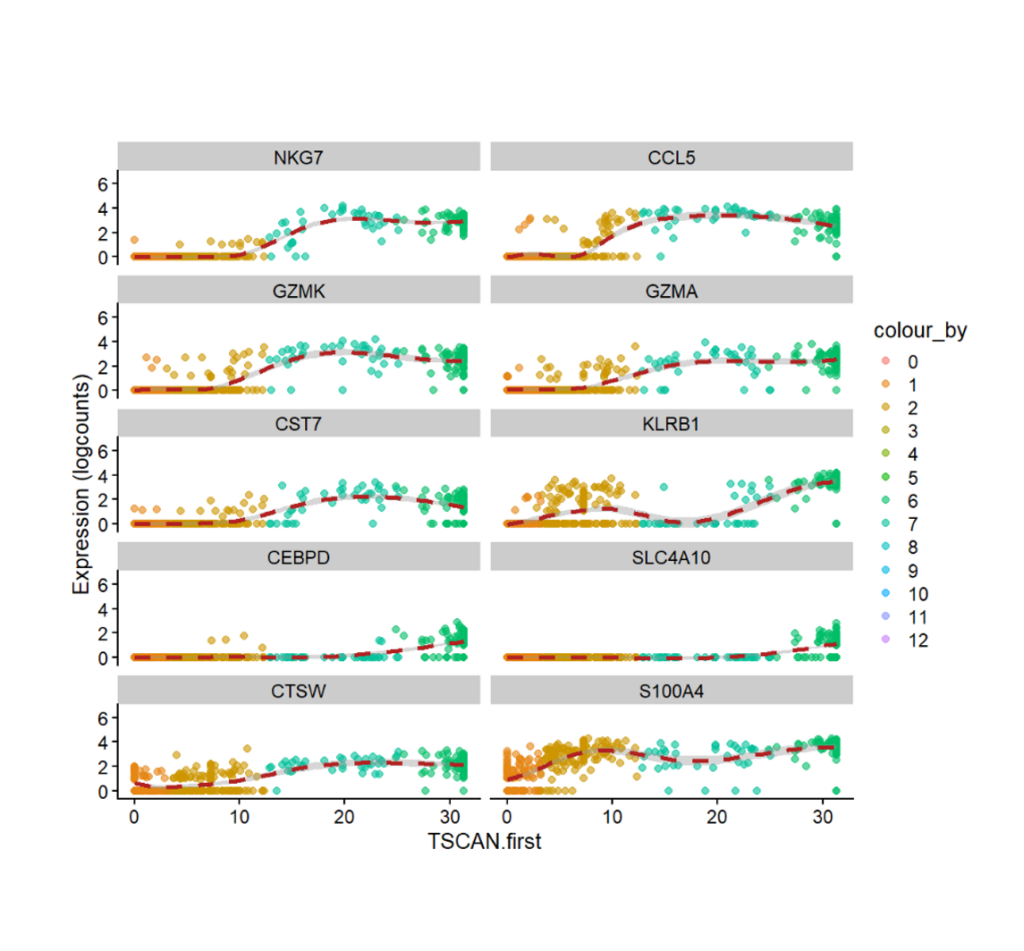
TOP 10 Expressed Genes (Up & Down) in a trajectory line
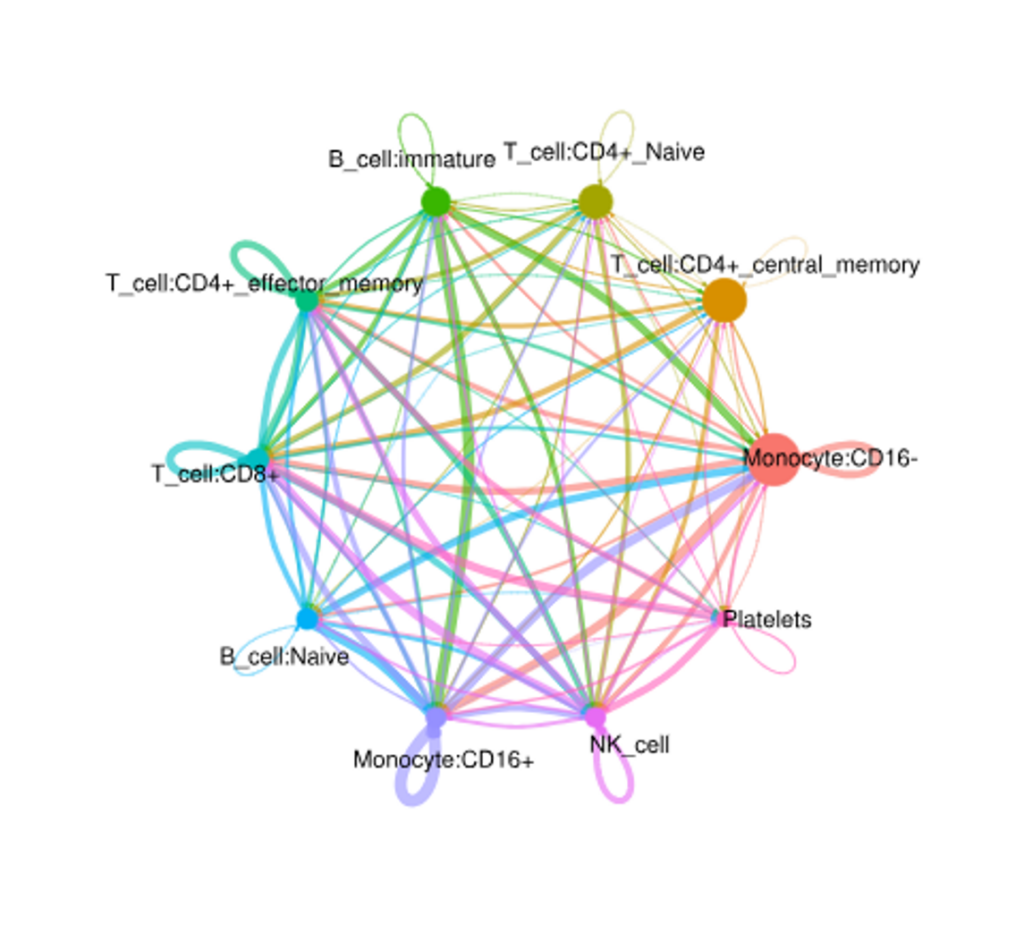
Cell chat
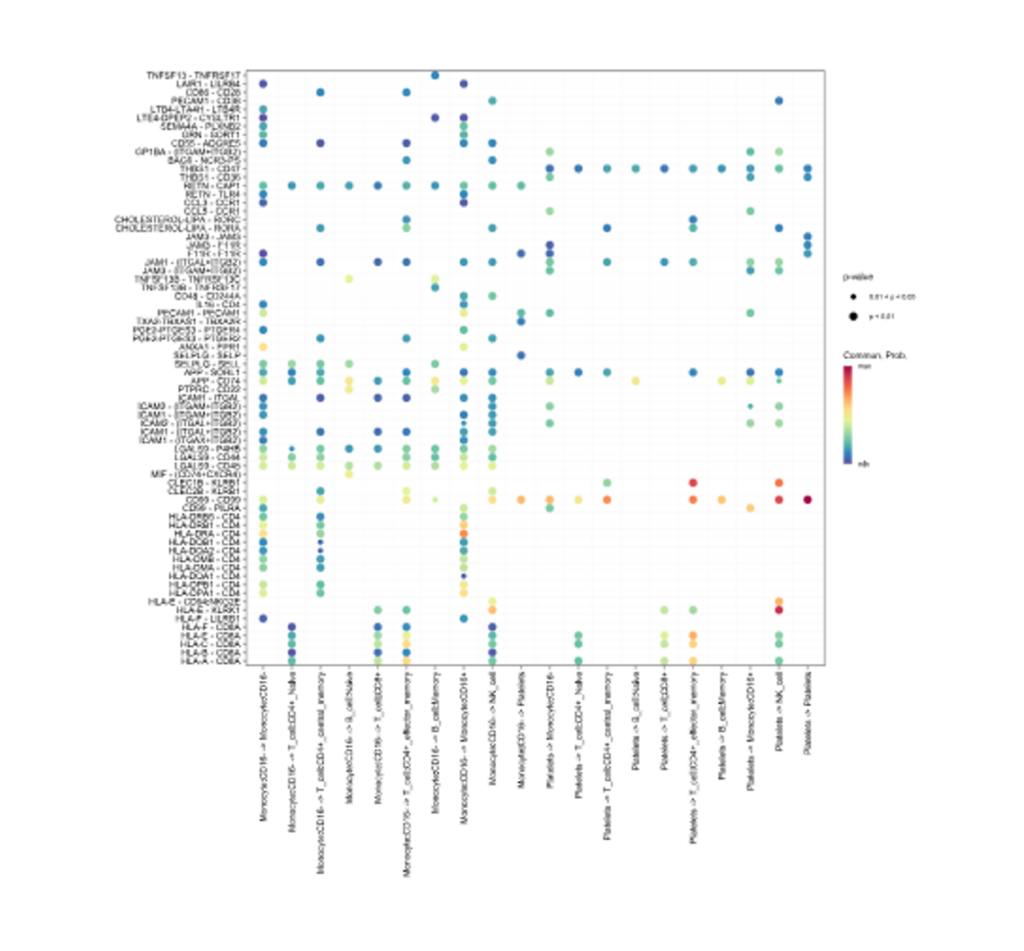
Heatmap cell chat
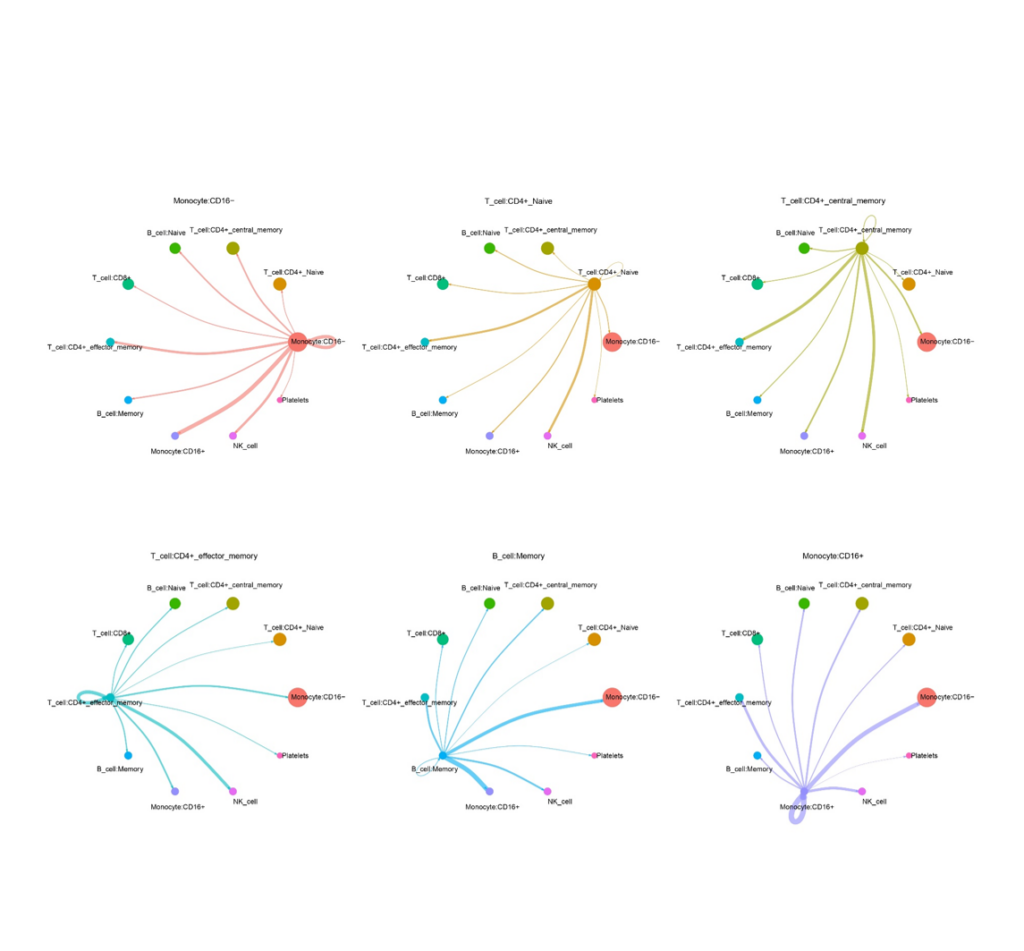
Principal Conections Between Cells
Opciones disponibles
Nos adaptamos a las necesidades de cada cliente a través de dos paquetes de análisis y la posibilidad de realizar un análisis completamente personalizado.
Paquete estándar
- Principal Component Analysis
- Cell Clustering (2D & 3D)
- Cluster Evaluation
- Differentially Expressed Genes
- TOP Differentially Expressed Genes
- RNA Velocity
- Pathways Enrichment (GSEA & GO Terms)
- Resultados en Genome One Reports
Paquete ampliado
- Todo lo incluído en el paquete estándar
- Trajectory Analysis
- Top 10 Expressed Genes (Up & Down) in a trajectory line
- Cell Chat
- Resultados en Genome One Reports


























