Análisis Transcriptómicos
La importancia de la expresión génica en el estudio de diferentes enfermedades
Portada » Transcriptómica
Estudio de la expresión génica
Los análisis transcriptómicos permiten cuantificar la expresión génica mediante el uso de plataformas NGS. La interpretación de los datos procedentes de la secuenciación del ARN (RNA-Seq) se ha convertido en un servicio cada vez más demandado debido a su sensibilidad y precisión.
El análisis bioinformático que llevamos a cabo en Dreamgenics incluye:
- Control de calidad de las secuencias.
- Alineamiento de lecturas frente a secuencias de referencia.
- Cuantificación de la expresión génica de la muestra.
- Análisis de la expresión génica diferencial con p-valor y con p-valor ajustado.
- Estudio de enriquecimiento de ontologías génicas y pathways.
- Estudio de isoformas generadas en eventos de splicing alternativo*.
- Identificación de otros RNAs (smallRNAs y ncRNA)*.
*Cuando el diseño experimental y la cobertura lo permitan.
Archivos de entrada
Podemos llevar a cabo el análisis bioinformático a partir de:
- Illumina: FASTQ
- MGI: FASTQ
Puedes enviarnos tus archivos crudos en un disco duro o compartirlos a través de un FTP.
Resultados en Genome One Reports
Entregamos los resultados de nuestros análisis transcriptómicos a través de nuestra plataforma Genome One Reports. Una plataforma web sencilla e intuitiva que te aportará las siguientes ventajas:
- Tendrás acceso al análisis de calidad de las secuencias obtenidas
- Análisis de correlación de muestras y estudio diferencial de expresión con p-valor y p-valor ajustado
- Gráficos PCA plot, Histogram, MA plot, Volcano plot, Heatmap y Correlation heatmap
- Enriquecimiento de rutas y términos Gene Ontologies (WikiPathways, GO-MF, GO-CC y GO-BP)
- Todas las tablas y figuras son fácilmente exportables, listas para ser utilizadas en publicaciones científicas
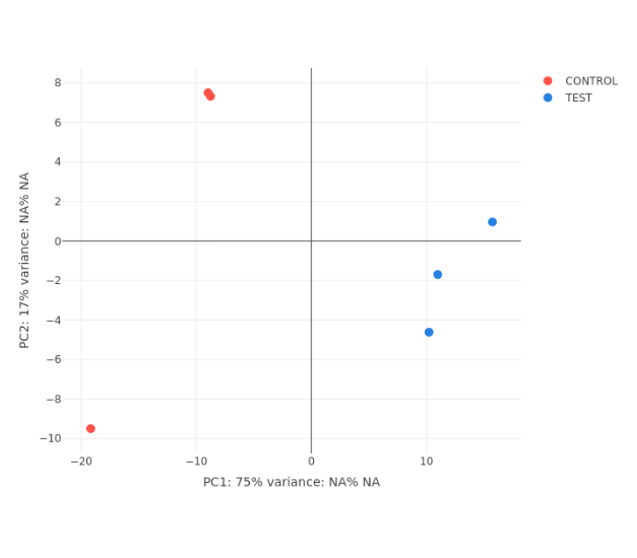
PCA Plot
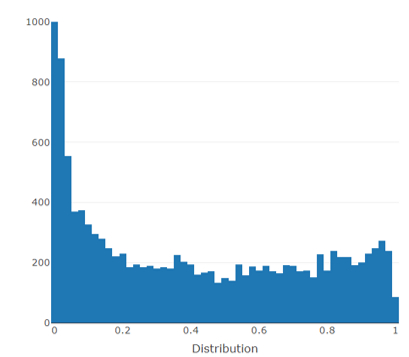
Histogram
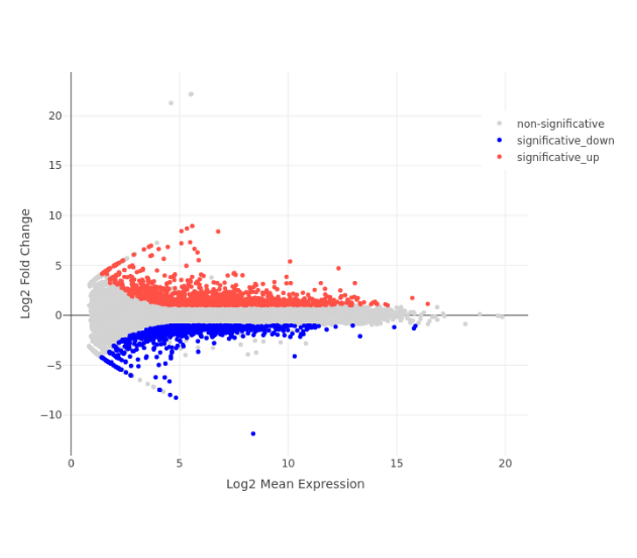
MA Plot
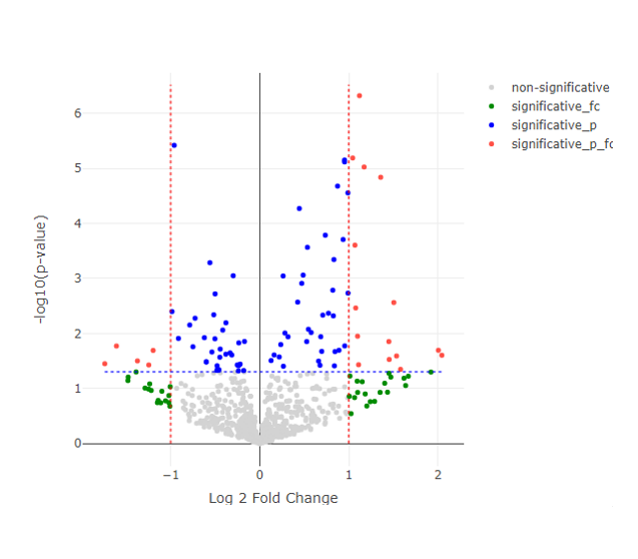
Volcano Plot
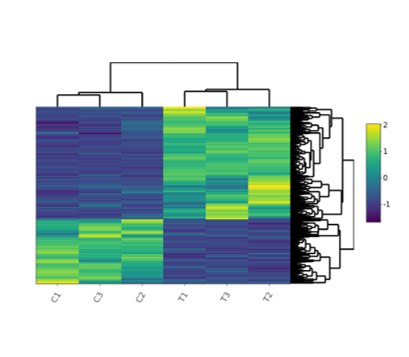
Heatmap
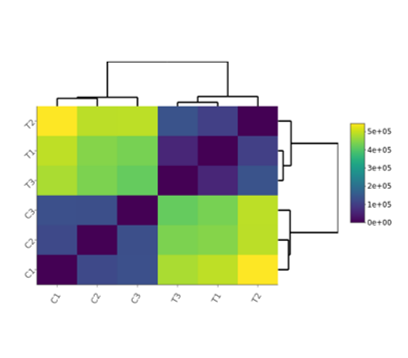
Correlation Heatmap
Diferentes opciones en experimentos RNA-Seq
Las librerías utilizadas en un experimento RNA-Seq depende de la muestra de partida y del tipo de ARNs que se quieran estudiar.
Contacta con nosotros y te ayudaremos a elegir la mejor opción para tu proyecto.
mRNA-Seq & Total RNA-Seq
- Plataforma DNBSEQ™-G400 (IVD)
- Librería MGIEasy Fast RNA Library
- Análisis de mRNA/mRNA + lncRNA
- >1ug de RNA con RIN>7
- Recomendado 40-60 M/muestra*
- Mínimo 3 muestras por condición
- Expresión diferencial, ontologías y pathways
- Resultados en Genome One Reports
miRNA-Seq
- Plataforma DNBSEQ™-G400 (IVD)
- Librería MGIEasy Small RNA Library
- Análisis de miRNA
- >1ug de RNA con RIN>7
- Recomendado 20 M/muestra
- Mínimo 3 muestras por condición
- Expresión diferencial, ontologías y pathways
- Resultados en Genome One Reports
*Eliminación de RNA ribosomal y mRNA de globinas en muestras de sangre para Total RNA-seq.


























